Analyse von DNA – der genetische Fingerabdruck
Das Restriktionsenzym Ban II schneidet.....
Schneiden des Plasmids pUCD-lacZ mit dem Restriktionsenzym Ban II
Bitte benutzen Sie die obigen Links zur Navigation, um zu Ihrer gewünschten Stelle zu springen, oder lesen Sie das ganze Protokoll indem Sie einfach runterscrollen.
Projekt des Biologie Leistungskurses des Marion-Dönhoff-Gymnasiums
Vom 23.01.-13.02.2007
Versuchsprotokoll
Zur
Analyse der DNA-
Schneiden des pUCD-lacZ mit dem
Restriktionsenzym Ban II
Abgabedatum: 01.03.2007
Protokollanten
Ann Katrin Cordes, Marisa Siemers, Daniel Beulcke, Leonie Wolf, Christin Paap und Katharina Kahrs
Inhaltsverzeichnis
Analyse von DNA – der genetische Fingerabdruck………………………………………...3
Das Restriktionsenzym Ban II schneidet an folgender DNA – Sequenz…………………..3
1A: Schneiden des Plasmids pUCD-lacZ mit dem Restriktionsenzym Ban II…………....4
- Vorbereitungen und Sicherheitsmaßnahmen
- Restriktionsansatz
1B:Elektrophorese und Auswertung…………………………………………………..…….5
- Herstellung des Elektrophoresepuffers
- Lösen der Agarose
- Gießen des Agarose-Gels
- Vorbereitung der Proben für die Elektrophorese
- Elektrophorese
- Vorbereitung der Elektrophoresekammer
- Auftragen der Proben auf das Agarose-Gel
- Start der Elektrophorese
- Färben der DNA im Agarose-Gel
- Verdünnen der Färbelösung (AzurB-Chlorid)
- Färben des Agarose-Gels
- Entfärben des Agarose-Gels
- Zu erwartenden Elektrophorese- Ergebnisse
- Versuchsergebnisse
- Auswertung
Quellenverzeichnis…..............................................................................................................13
Analyse von DNA –
der genetische Fingerabdruck
Einleitung
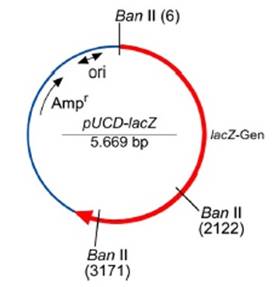
Die Anfänge der DNA–Analyse liegen in den 70er Jahren. Damals wurden die Restriktionsenzyme erstmals entdeckt, die zum Schneiden eines Plasmids, ringförmiger DNA, benötigt werden. Schneidet man ein Plasmid, so entstehen so genannte „sticky-ends“ (klebrige Enden). Jedes Restriktionsenzym schneidet an einer anderen Stelle des Plasmids und hat dementsprechend andere „sticky-ends“. Für unseren Versuch haben wir das Restriktionsenzym Ban II verwendet, dass an folgenden Stellen schneidet:

Abb.: Schematische Darstellung des Plasmids pUCD-lacZ (Quelle: Bio-Rad/Blue Genes)
Das Restriktionsenzym Ban II schneidet an
folgender DNA – Sequenz:
5`… GRGYC …3` *
3`… CYGRG …5`
*Y = C oder T; R = G oder A
Nach dem Schneiden des Plasmids wird eine Gel-Elektrophorese gefahren, bei der die DNA-Fragmente getrennt werden. Ziel der Gel-Elektrophorese ist der Nachweis, dass die DNA wirklich geschnitten wurde.
Für den Versuch benötigt man sehr viel DNA, die wir nicht selber vervielfältigen mussten, da sie uns zur Verfügung gestellt wurde.
Die Analyse von DNA
ist während der letzten Jahre in verschiedenen Gebieten immer wichtiger
geworden. Bei der Aufklärung von Verbrechen ermöglicht sie durch
DNA-Identifizierungsmuster die Überführung des Täters, z.B. durch ein einziges
Haar. Auch Verbrechen, die bereits viele Jahre zurückliegen, können heute mit
Hilfe der DNA – Analyse aufgeklärt werden. Diese Analyse wird auch als
genetischer Fingerabdruck bezeichnet. Ein weiteres Einsatzgebiet der DNA –
Analyse ist die Ermittlung von Vaterschaften, die durch Blutuntersuchungen nicht
sicher nachgewiesen werden können.
1A: Schneiden des Plasmids pUCD-lacZ mit dem Restriktionsenzym Ban II
Vorbereitungen und Sicherheitsmaßnahmen:
Um ein optimales Ergebnis der Versuche zu erhalten, ist es wichtig, dass folgende Sicherheitsmaßnahmen im „Labor“ eingehalten werden:
Steriler Arbeitsplatz an dem nicht gegessen und getrunken werden darf
Schutzkittel tragen
Beim Pipettieren werden grundsätzlich Handschuhe getragen
Alle Reagenzien müssen vor Gebrauch vollständig aufgetaut sein und durchmischt werden. Hierzu werden die Gefäße leicht angeschnipst
Noch leicht gefrorene Reagenzien werden schonend mit den Händen aufgetaut
Anschließend werden die Reagenzien umgehend wieder auf Eis gelegt. Dabei ist zu beachten, dass das Restriktionsenzym durch den Zusatz von Glycerin nicht aufgetaut werden muss, die Enzyme und Nukleinsäuren allerdings immer auf Eis stehen müssen
Jeglichen Kontakt mit den Pipettenspitzen vermeiden
Bevor man das Pipettieren beginnt, muss die gesamte Reagenzienmenge in der Spitze des Eppendorfgefäßes gesammelt werden, was entweder durch Zentrifugieren oder durch Anschnipsen erreicht werden kann
Benutzte Materialien werden ordnungsgemäß entsorgt
Die hinter den Reagenzien angegeben Röhrchen bezeichnen die von Blue Genes vorgegebenen Reagenzien. Diese sind zur besseren und schnelleren Erkennung mit Nummern bezeichnet.
Restriktionsansatz:
Reagenzien:
- 12 µl steriles Wasser (Röhrchen 14);
- 2 µl Puffer B (Röhrchen 4);
- 4 µl pUCD-lacZ (Röhrchen 11);
- 2 µl Ban II (Röhrchen 2)
Zudem benötigen wir ein Eppendorfgefäß, eine Eppendorfpipette (2-20µl), Pipettenspitzen, einen Behälter mit 37° C warmen Wasser und ein Styroporgefäß mit Eis.
Durchführung:
Zuerst wird ein Eppendorfgefäß für den Restriktionsansatz mit einem ‚R’ gekennzeichnet. Danach werden entsprechend der oben angegebenen Reihenfolge die Reagenzien in das vorbereitete Eppendorfgefäß pipettiert (für jeden Pipettierschritt jeweils eine neue Spitze, um Verunreinigungen zu vermeiden).
Durch Auf- und Abpipettieren wird der Restriktionsansatz gemischt. Anschließend wird der Restriktionsansatz durch Zentrifugation in der Spitze des Eppendorfgefäßes gesammelt. Der Restriktionsansatz wird nun bei 37° C etwa 30 Minuten inkubiert, damit das Restriktionsenzym Ban II das pUCD-lacZ schneiden kann. Dann wird die Probe bis zur weiteren Verwendung auf Eis aufbewahrt oder bei -20 ° C eingefroren.
1B: Elektrophorese und Auswertung
Herstellung des Elektrophoresepuffers:
Reagenzien:
- 30 ml TBE (10-fach konzentriert)
- 270 ml destilliertes Wasser
Man benötigt einen Erlenmeyerkolben, Messzylinder.
Durchführung:
30 ml des Elektrophoresepuffers (TBE) werden abgemessen und mit 270 ml destilliertem Wasser verdünnt.
Färben der DNA im Agarose-Gel:
Reagenzien:
- 30 ml 1-fach konzentrierter TBE-Puffer
- 300 mg Agarose
Zusätzlich benötigt man Messzylinder, einen Erlenmeyerkolben, eine Mikrowelle und eine Waage.
Durchführung:
300 mg Agarose werden abgewogen und zu 30 ml 1-fach konzentrierten Elektrophoresepuffer in dem Erlenmeyerkolben hinzu gegeben. Die Agarose wird mithilfe eines Mikrowellenherds gelöst (durch aufkochen). Zum Durchmischen der Lösung schwenkt man das Gefäß mehrmals. Anschließend lässt man die Lösung auf ca. 50 °C abkühlen.
Gießen des Agarose-Gels:
Außer dem Agarose-Gel benötigt man die Elektrophoresekammer, den Gelträger, die Kämme und die Trennkeile.
Durchführung:
Der Gelträger wird in vorgesehener Richtung in die Elektrophoresekammer gelegt, sodass die Kämme später korrekt eingesetzt werden können. Die Enden des Gelträgers werden durch einsetzen von Trennkeilen abgedichtet. Dann wird das Gel (die Agarose-Lösung) ca. 7 mm hoch auf den Gelträger gegossen. Die Probekämme werden kurz darauf an den hierfür vorgesehenen Stellen eingesetzt. Das Agarose-Gel wird ungefähr 20 Minuten bei Raumtemperatur gekühlt, bis es erstarrt. Während dieser Zeit darf die Kammer nicht bewegt werden. Ist das Gel erstarrt, werden die Trennkeile sowie die Kämme entnommen. Durch das Entfernen der Kämme entstehen kleine Kammern in die zu späterem Zeitpunkt die Reagenzien eingefüllt werden können.
Vorbereitung der Proben für die Elektrophorese
DNA-Marker:
Reagenzien:
- 8 µl DNA-Marker (Röhrchen 8)
- 12 µl steriles Wasser (Röhrchen 14)
- 2 µl DNA-Auftragspuffer
Zusätzlich benötigt man ein Eppendorfgefäß, eine Pipette (2-20 µl) und Pipettenspitzen.
Durchführung:
Zuerst wird das Eppendorfgefäß für den DNA-Marker mit einer ‚1’ markiert. Dann werden die Reagenzien in oben angegebener Reihenfolge in das beschriftete Gefäß pipettiert. Wieder wird die Flüssigkeit durch Schnipsen oder Zentrifugieren am Gefäßboden gesammelt und sofort wieder auf Eis gestellt.
Kontrolle
Reagenzien:
- 4 µl ungeschnittenes Plasmid pUCD-lacZ (Röhrchen 11)
- 16 µl steriles Wasser (Röhrchen 14)
- 2 µl DNA-Auftragspuffer
Zusätzlich benötigt man ein Eppendorfgefäß, eine Pipette (2-20 µl) und Pipettenspitzen.
Durchführung:
Das Eppendorfgefäß für die Kontrolle wird mit einer ‚2’ markiert. Dann werden alle Reagenzien in oben angegebener Reihenfolge in das beschriftete Gefäß pipettiert. Die Flüssigkeit wird durch Schnipsen oder Zentrifugieren am Gefäßboden gesammelt und sofort wieder auf Eis gestellt.
Restriktionsansatz:
Man benötigt den zu Beginn gefertigten Restriktionsansatz (20 µl), 2µl DNA-Auftragspuffer, ein Eppendorgefäß, eine Pipette (2-20 µl) und Pipettenspitzen.
Durchführung:
Zu dem bereits angesetzten Restriktionsansatz werden 2 µl DNA-Auftragspuffer hinzu pippetiert. Die Flüssigkeit wird durch Schnipsen oder Zentrifugieren am Gefäßboden gesammelt und sofort wieder auf Eis gestellt.
Elektrophorese
Reagenzien:
- Elektrophoresepuffer
- Jeweils 10 µl des DNA-Markers, der Kontrolle und des Restriktionsansatzes Ban II
Man benötigt die Elektrophoresekammer, Pipetten (2-20 µl) , Pipettenspitzen, eine Spannungsquelle und zwei Kabel.
Vorbereitung der Elektrophoresekammer:

Die Elektrophoresekammer wird so aufgestellt, dass der negative Pol (schwarz) links und der positive Pol (rot) rechts zu finden ist. Es wird so viel Pufferlösung in die Kammer gegeben, dass das Gel 1 mm überdeckt wird.
Auftragen der Proben auf das Agarose-Gel
Das Pipettieren der Proben in die Geltaschen erfolgt, nachdem das Gel mit dem Puffer überdeck wurde. Die oben angegebenen Proben müssen in der richtigen Reihenfolge und sehr langsam und vorsichtig in die Taschen eingefüllt werden.
Jede Gruppe pipettiert ihre 3 Probelösungen immer in der angegebenen Reihenfolge:
Spur 1: 10 µl DNA-Marker (1)
Spur 2: 10 µl Kontrolle (2)
Spur 3: 10 µl Restriktionsansatz Ban II (R)
Start der Elektrophorese:
Nachdem alle Proben in die Taschen pipettiert wurden, wird die Elektrophoresekammer mit dem Deckel geschlossen. Durch die Kabel wird jener an eine Spannungsquelle angeschlossen, welche auf 100 V eingestellt wird. Die Elektrophorese nimmt mindestens 20 Minuten in Anspruch, besser sind jedoch 30 Minuten. Bevor der blaue Farbstoff die zweite Geltaschenreihe erreicht, muss die Elektrophorese beendet werden.
Durch den hinzu gegebenen Ladepuffer entsteht ein Überschuss an OH- -Ionen. Die durch die Abgabe von Protonen negativ geladene DNA, wird von der Anode angezogen. Daher wandern die Elektronen der DNA-Fragmente von dem (-)-Pol zum (+)-Pol. Das Agarose-Gel besitzt eine Netzstruktur. Diese dient dazu, dass die DNA-Fragmente unterschiedlich weit wandern. Fragmente mit weniger Basenpaaren wandern weiter, als die mit mehr Basenpaaren. Dieses liegt an der größeren Reibung. Die DNA wird also in dem Gel nach ihrer Basenlänge getrennt. Die Basenpaarlänge wird durch die Spur des DNA-Markers bestimmt, da diese die Längenvorgaben gibt und zeigt, welche Bande für welche Länge des DNA-Fragments steht. Die unterschiedlichen Fragmente des Markers stehen für Längen, die bereits alle bekannt sind. So kann man der 1. Bande die Basenlänge 21226, der 2. Bande die Läge 4796 zuordnen.
Färben der DNA im Agarose-Gel:
Verdünnen der Färbelösung (AzurB-Chlorid)
Reagenzien:
- 500 ml destilliertes Wasser
- 1 ml AzurB-Chlorid (Röhrchen 15)
Zusätzlich benötigt man einen Erlenmeyerkolben, eine Pipette (200-1000 µl) und Pipettenspitzen.
Durchführung:
Die oben genannten Komponenten werden gemischt und bei Raumtemperatur in einer dunklen Flasche aufbewahrt. Die Lösung kann mehrmals verwendet werden.
Färben des Agarose-Gels:
Die Färbelösung wird in eine Petrischale gegeben, anschließend wird das Agarose-Gel vorsichtig dazugelegt, welches 15 Minuten in der Färbelösung verbleibt. Die Färbelösung kann nach der Verwendung wieder in die Flasche gegeben und nochmals verwendet werden.
Entfärben des Agarose-Gels
Das Gel wird in einer Schale mehrmals mit destilliertem Wasser gespült. Hierdurch wir es entfärbt und die Bandenmuster werden nach ungefähr 10 Minuten gut erkennbar sein.
Zu erwartende Elektrophorese-Ergebnisse:
Nach der Durchführung der Elektrophorese sollten in den einzelnen Spuren der drei Taschen mit unterschiedlich gefüllten Substanzen Banden zu erkennen sein.
In der ersten Spur mit dem DNA-Marker sollten im Idealfall 12 Banden zu erkennen sein. Diesen kann man dann die unterschiedlichen Fragmentgrößen zuordnen (siehe Arbeitsblatt 6).
Eine, maximal zwei, Banden kann man in Spur zwei erkennen, in der die Kontrollsubstanz enthalten ist mit dem ungeschnittenen pUCD-lacZ. Zum einen bei etwa 5669 bp, diese Länge entspricht einer pUCD-lacZ-DNA und zum zweiten eine von einer Spezialform dieses Plasmids, Supercoil-Form. Diese Form ermöglicht dem Plasmid leichter durch die Netzstruktur des Agarose-Gels zu wandern als in der normalen Ringform. Diese Form sollte demnach weiter gewandert sein als die Ringform des Plasmids.
In der dritten Spur müssten drei Banden zu sehen sein, da das Restriktionsenzym BanII das pUCD-lacZ Plasmid an zwei Stellen schneidet und so drei verschieden große DNA-Fragmente vorliegen, wenn die Trennung funktioniert hat. Zu erwarten sind die Banden bei 1049 bp, 2116 bp und 2504 bp.
Funktioniert hat das Schneiden des Plasmids pUCD-lacZ, wenn die Erwartungen erfüllt werden.
Spur 1: 10 μl geschnittenen pUCD-lacZ-Plasmid
Spur 2: 10 μl ungeschnittene Plasmid pUCD-lacZ
Spur 3: 10 μl DNA-Marker
Nach Beendung der Elektrophorese, waren in der Spur des DNA-Markers (Spur 3) acht Banden zu erkennen, sechs davon ungefähr gleichgroß, zwei allerdings deutlich breiter. Da man eigentlich 12 Banden hätte erkennen sollen, kann man daraus schließen, dass die beiden breiteren Banden sich aus mehreren Banden zusammensetzen müssen. Die erste Bande, 7,5 mm von der Tasche entfernt, kann man dem mit Abstand größten DNA-Fragment zuordnen, welches aus 21226 bp (Basenpaaren) besteht. Nach einer Wanderungsstrecke von 11,5 mm folgt dann eine recht breite Bande mit einer Länge von 4796 bp. Da sie relativ breit ist, kann man schließen, dass sie sich aus drei sehr dicht beieinander liegenden Fragmentgrößen (5148 bp; 4973 bp und 4268 bp) zusammensetzt. Der Mittelwert dieser drei Größen ergibt dann 4796 bp.
1 mm weiter folgt eine weitere Bande, nicht ungewöhnlich breit, deswegen wird dieser die Fragmentgröße 3530 bp zugeordnet. Nach insgesamt 17 mm Wanderungsstrecke folgt eine weitere, breite Bande. Da die folgenden Fragmente nur einen Größenunterschied von 123 bp aufweisen, ist es nahe liegend, dass jene nur als eine Bande zu erkennen sind, diese wird also den Fragment-größen 2027 bp und 1904 bp zugeordnet. Die nächsten vier Banden sind sehr deutlich zu erkennen, sodass man ihnen ohne weiteres die Fragmentgrößen 1584 bp, 1375 bp, 947 bp und 831 bp zuordnen kann. Die letzte Bande, die einer Fragmentgröße von 564 bp zuzuordnen gewesen wäre, ist auf dem Gel nicht mehr zu erkennen. So kommen wir letztendlich auf die 12 Banden, die laut Literatur in der dritten Spur des DNA-Markers zu erwarten waren.
In der zweiten Spur wurde während der Elektrophorese das ungeschnittene Plasmid pUCD-lacZ gefahren. Hier erkennt man sofort das zu erwartende Ergebnis von zwei Banden. Die erste entspricht der Fragmentgröße des Plasmids von ca. 5669 bp, die zweite ist auf die erwartete Spezialform des Plasmids zurückzuführen, der Supercoil-Form.
In der ersten und letzten Spur mit dem geschnittenen pUCD-lacZ-Plasmid, sind drei Banden zu erkennen, den unterschiedlichen Fragmentgrößen entsprechend. Die Werte werden mathematisch analysiert, um zu überprüfen, ob sie den Literaturwerten entsprechen.
Auswertung:
"Die zurückgelegte Wanderungsstrecke der DNA-Fragmente im Agarose Gel ist umgekehrt proportional zum Logarithmus (log) des Molekulargewichtes (M) der Fragmente. Das Molekulargewicht eines bestimmten DNA-Fragmentes ist proportional zur Anzahl der Basenpaare bp" (Arbeitsblatt 6). Dies ist als Zusammenhang zwischen Wanderungsstrecke bzw. Molekulargewicht eines bestimmten DNA-Fragments und tatsächlicher Fragmentgröße in bp gegeben.
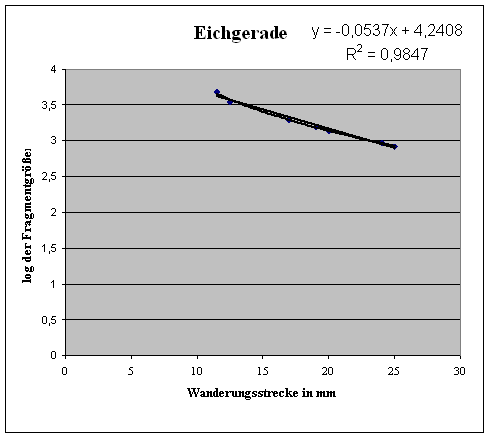
In Excel wird daraufhin eine Wertetabelle erstellt, die die gemessene Wanderungsstrecke mit dem Logarithmus des zugehörigen Literaturwerts der Fragmentgrößen in Verbindung bringt. (Abb.1) Daraufhin wird ein Punktdiagramm erstellt, in welches nach Vernachlässigung der Fragmentgröße 21226 bp eine Eichgerade berechnet wird. Das Fragment wurde nicht berücksichtigt, da seine Größe stark von der untersuchten Fragmentgröße abweicht (Abb.2).
Anhand der vom Computer berechneten Funktion dieser Eichgeraden kann man nun die Wanderungsstrecke der drei Banden des Restriktionsansatzes in den Logarithmus der Fragmentgröße umrechnen. Diesen Wert muss man mit 10 potenzieren um die eigentliche Fragmentgröße in Basenpaaren zu erhalten (Abb.3).
|
Fragmentgröße in bp |
log der Fragmentgröße |
Wanderungsstrecke in mm |
|
4796 |
3,680879174 |
11,5 |
|
3530 |
3,547774705 |
12,5 |
|
1966 |
3,293583513 |
17 |
|
1584 |
3,199755177 |
19 |
|
1375 |
3,138302698 |
20 |
|
947 |
2,976349979 |
24 |
|
831 |
2,919601024 |
25 |
(Abb. 1)
(Abb. 2)
|
Wanderungs-strecke |
Log |
Fragmentgröße |
Zu erwartende Fragmentgröße (Blue Genes) |
Prozentuale Abweichung |
|
16 |
3,3816 |
2408 |
2504 |
4,0 |
|
17 |
3,3279 |
2128 |
2116 |
0,6 |
|
22 |
3,0595 |
1147 |
1049 |
9,3 |
(Abb. 3)
Unsere zu erwarteten Ergebnisse haben sich mit Hilfe der Elektrophorese bestätigt. Wie bereits in der Auswertung erwähnt, haben uns alle drei Spuren eindeutige und positive Ergebnisse gezeigt. Unsere erhaltenden Fragmentgrößen haben auch nur sehr geringe Abweichungen zu den Literaturwerten von Blue Genes. Die Abweichungen liegen zwischen 0,6 % und 9,3 %. Diese Abweichungen lassen sich beispielsweise durch Ungenauigkeiten in der Durchführung des Versuches oder bei der Abmessung der Wanderungsstrecken erklären. Dennoch sind die Abweichungen relativ gering und somit ist unser Ergebnis sehr zufriedenstellend.
Dieser Versuch diente als Grundlage und Einführung für weitere Versuche zur Klonierung von DNA.
Insgesamt kann man sagen, dass der Versuch sehr erfolgreich war und wir durch ihn praktische Grundlagen zur Gentechnik erfahren haben.
BioRad; Blue Genes; Analyse von DNA, Schneiden des pUCD-lacZ mit dem Restriktionsenzym BanII, Arbeitsblatt 1-6
http://www.sg.hdh.bw.schule.de/neu/bluegene.htm
Protokollanten
Ann Katrin Cordes, Marisa Siemers, Daniel Beulcke, Leonie Wolf, Christin Paap und Katharina Kahrs